生物医学语义关系抽取(SemanticRelation Extractions, SRE)是对疾病、药物、蛋白质、基因等重要医学实体之间语义关系 (如治疗关系、诱导关系、突变关系)的揭示,是构建领域知识图谱、本体与知识库、临床决策支持系统的重要基础,以进步辅助智慧医疗与精准医学,具有重要的现实意义。在理论研究上,关系抽取属于信息抽取(Information Extraction, IE)的一部分,Culotta等对它的定义是“发现实体之间的语义关系,,包括两个步骤:(1)确定兴趣实体对之间是否表示了一定关系;(2)将检测到的关系分类到预定义的类别中。
在过去数十年,生物医学SRE得到深入研究,在方法上取得了巨大进步,并目一有一些较成熟的平台和土具免费对公众开放,如美国国立医学图书馆(NLM)的SemRe等。这些方法与土具主要是将医学领域良好的知识组织系统(主题词表、本体、一体化医学语言系统等),与自然语言处理(NLP技术、人工定义的规则和总结的特征等相结合,并取得了一定的成效。
近年来,在大数据浪潮和大规模计算的推动下,深度学习(Deep Learning)方法重新复苏、并焕发出勃勃生机。其中,卷积神经网络(Convolutional Neural Networks,CNNs不但在图像分类和语音识别上作出了巨大的贡献,而目一在NLP任务、特别是文本和关系分类方面,也取得了良好效果。CNNs使用一定维数的词向量来表示基本单元(tokens(单词、字母等),实现了语义知识表示的数字化和可计算性,是NLP具有一定革命意义的突破。生物医学是个既有良好知识资源又对语义关系抽取有极大现实需求的领域,同时,也存在着较高的专业壁垒。如何将深度学习方法融入医学领域,是一个值得思考的问题。全面系统的综述将会对前人研究进行彻底梳理和消化吸收,从而为深度学习在生物医学领域的探索提供启示。
1研究方法和过程
本研究参考美国医学研究所制定的《系统综述研究标准》,具体实现方法和过程如下:
1.1数据源和检索策略
对PubMed, Web of Science和IEEE三个数据库进行检索,时间跨度为2000年1月一2016年12月。检索策略为与SRE相关的关键词,如“relation extraction", "semanticrelation", "drug-drug interaction"等,并不断进行调整与验证。同时,检索了涉及关系抽取内容的重要生物医学评测网站,包括BioCreative, i2b2, SemEval等,以便全面覆盖生物医学SRE文献。
1.2研究遴选
检索共得到16827篇题录信息,去重后为11714篇,并进一步排除非生物医学领域、与语义关系抽取无关、非英语等方面的文献,筛选出相关性较好的856篇。为找到最典型SRE方法,进步遴选代表性研究。遴选标准包括在同类研究中取得最好效果、或第一次提出方法、或来自相对权威的研究机构和期刊等,最后确立对125篇进行全文下载和详细分析。
1.3数据分析
对全文内容进行深度分析,从研究目的、力一法、语料与实现效果四个维度展开。其中,SRE方法是分析重点,包括其基本原理、优化思路和实现算法,以便通过原理层的解析和归纳,将生物医学SRE方法的探索历程和研究范式进行科学的阶段划分。
2研究结果
Etzioni等(2008)将关系抽取方法分为三类:基于知识、有监督和无(自)监督。 Zhou等(2014)将其分为两类:基于知识(依赖资源和预定义模式)和机器学习 (基于良好定义的特征)。最近五年,深深度学习方法在NLP领域展示巨大潜力。Liu等(2016)报道了使用CNNs进行药物一药物相互作用(Drug-Drug Interactions,DDI)关系抽取,在没有大量人工定义特征的前提下,实现了良好的效果[yob,预示着生物医学SRE研究的新范式。因此,结合文献调研,本研究将生物医学SRE方法分为三阶段:基于知识、传统机器学习与最新的深度学习,如下图1所示。从N-gram论元的角度,生物医学的语义关系包括二元和多元关系,本研究主要关注二元关系。
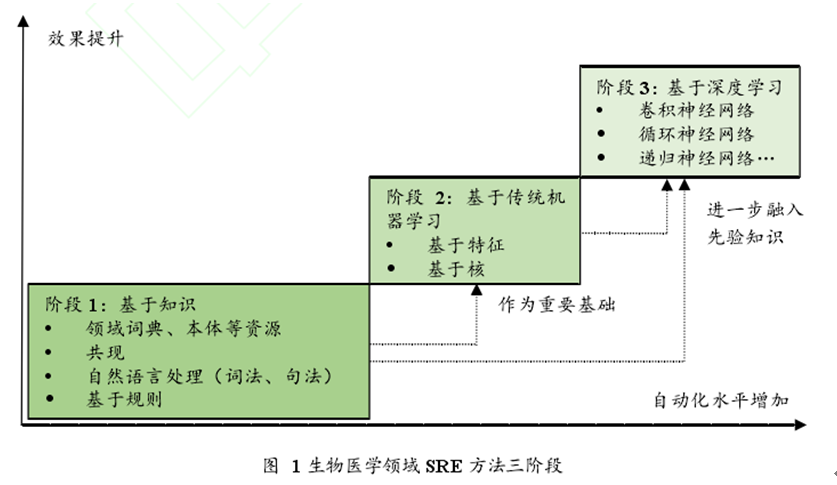
2.1基于知识的方法
基于知识的SRE方法主要包括:(1)借助医学知识资源(叙词表、本体等),并与(2)共现分析、(3)符号自然语言处理 (词法、句法分析等)、(4)人工总结规则 (模板/模式匹配)等相结合。
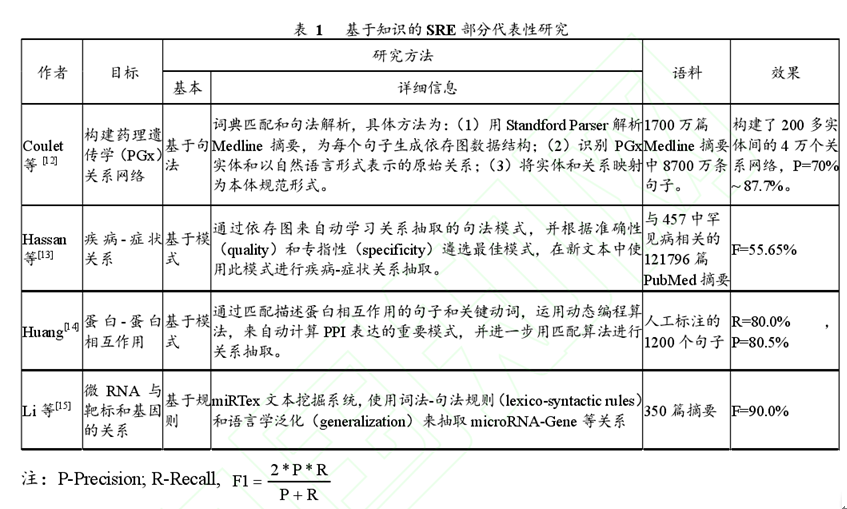
基于知识的SRE主要基于论元一谓词范式(argument-predicate paradigm,语义谓项(主一谓一宾三元组)识别是关系抽取的核心部分。因此,谓项黄金标准(predicationgold standard)的构建,谓词(关系触发词)的识别和分类,是研究者关注的重点。如美国国立医学图书馆(NLM)的Kilicoglu等(20ll)构建了语义谓项金标准,并对影响Caffaots、相关(associated with等数十个谓词进行了基于本体层面的定义与示例说明。NLM开发的SemRep平台,是一个融合词典、语法和规则的语义解释器,支持从自由文本中抽取语义谓项。更多代表性研究请参见表1。
在具有良好词典资源、规则得到精细构建的生物医学子领域,基于知识的SRE方法取得了较好的效果。然而,由于自然语言表达的复杂多样性,基于人工总结的规则和模板常常难以覆盖各种语言现象,目一领域可移植性不佳。随着信息技术的发展,研究者们转向机器学习方法,在语料的支持下,运用统计学或判别式算法进行自动关系抽取与效果优化。
2.2 传统机器学习方法
机器学习方法将关系抽取视为一个分类问题[#]。对句子S=W1, W2 , ..., e1 ,..., wj ,...,e2 ,..., Wn,其中,Wi表示单词,e1, e2分别表示目标实体,分类函数f(.)表示如下:

T (S)表示从句子S中抽取的特征。如果有标注语料进行训练,函数f(.)可构建为一个判别式分类器(DiscriminativeClassifier,如感知机(Perception)或支持向量机(Support Vector Machines, SVMs),以判断句子中两个实体之间是否具有某种关系。
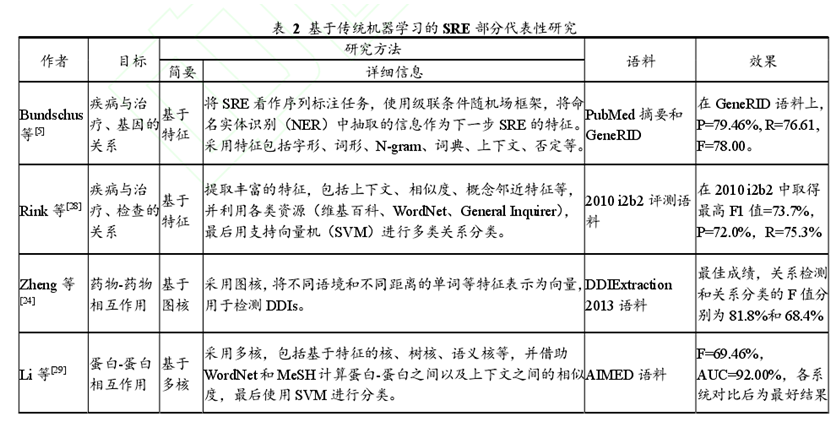
在生物医学SRE中,机器学习以有监督学习为主流,本研究也主要关注有监督方法。根据标注数据集中样本在模型中的表示力一式,关系抽取的有监督学习方法可进步分为:(1)基于特征的方法(Eeatare-based和(2)基于核的方法(Kernel-based)。其共同特点是利用语言学分析和知识资源将关系指称(relation mention转化为丰富的表示。与基于知识的方法不同的是,机器学习方法主要将上下文语境作为特征,而不是谓词信息。
2.2.1 基于特征的方法
基于特征的方法是从文本中生成句法和语义特征,以特征向量的形式呈递给分类器。句法特征常常包括两个实体、词性标签、词间顺序等;语义特征常常体现于依存分析 ( dependency parsing)中两个实体的路径。 Miwa等(2010)使用个丰富特征集合,对多个解析器(parser)结果进行构建,以抽取蛋白一蛋白相互作用(protein-proteininteraction, PPI,并获得了所有评测系统中的最好效果。 Xu 02016)等将基于特征的机器学习算法与领域知识融合,构建化合物诱导疾病关系(chemical-induceddisease relations, CID)抽取的端到端系统。该系统由命名实体识别和关系抽取两部分组成:(1)疾病和化合物命名实体识别(CRF算法)、正则化模块(采用向量空间模型);(2)化合物一诱导疾病关系抽取模块,用SVM进行结果分类,在BioCreative V CDR评测中取得最佳成绩。
关系抽取的自然语言处理,需要将输入数据进行结构化表示以提取特征,而获取特征的最优子集常存在困难。因此,为了解决遴选合适特征集合的问题,研究者设计了专门的核(kernels,以实现输入数据的丰富表示。
2.2.2 基于核的方法
基于核的方法将实体关系对编码为某种结构,如序列、树、图、依存关系路径等形式;而核被定义为计算对象之间相似度的函数。根据实体关系为被编码对象选择合适的核是关键。核方法的优势是在多项式计算时提供大的特征空间,而无需明确表示这些特征。
Lodhi等(2002)首次使用线性核(stringkernels)进行关系抽取}zy。如有两个线性序列x和y,线性核计算两个序列中子序列的才刚以度。子序列相似度越高,则两个序列的相似度则越大。Sun等(2015)设计了一个高效和可扩展系统,使用线性核来识别DDI信息。结果表明,当有丰富的词法和句法特征集合时,线性SVM分类器可实现具有竟争性的效果。此外,也有其他核进行句子和句法结构相似度计算的报道,如树核(tree kernel),图核(graph kernel)、子序列核(subsequence kernel )等,或者是多种核的组合。基于特征和基于核的SRE代表性研究请见表2。
生物医学SRE的传统机器学习方法主要基于抽取的特征或设计的核,在训练语料支持下,一定程度实现自动化和较好性能;不足之处是特征土程较为复杂,依赖NLP技术进行句法分析(如依存路径解析)等处理,容易衍生错误、造成错误级联传播。
2.3 深度学习方法
深度学习致力于从数据中自动学习更 一般的从底层特征到高层概念的多层抽象表示,成为近年来人工智能和NLP的研究热点。以卷积神经网络(CNNs)为代表深度学习方法,运用到各种NLP任务中,特别是句子和关系分类,在没有复杂的手土定义特征的前提下,取得了良好的效果。
2.3.1 CNNs基本原理
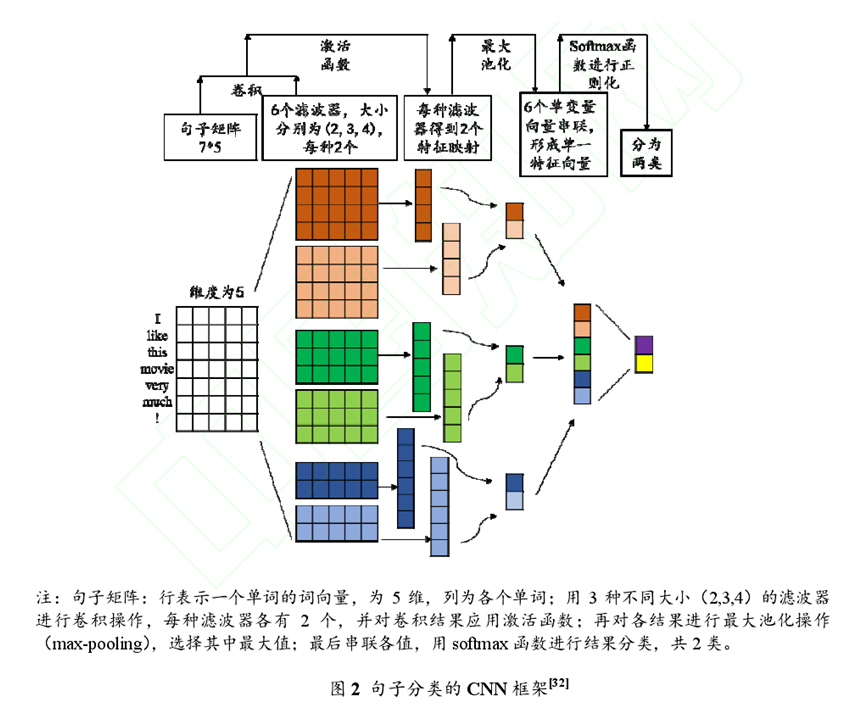
CNNs是一种前馈型人工神经网络,最初应用于图像分类,核心特点是局部感知和权值共享。实现机制为:对输入层使用不同的滤波器(filters)进行卷积操作,对每层结果应用非线性激活函数(ReLU或tank再进行池化(pooling,最终再将结果组合用softmax进行分类。在NLP应用中,输入值是句子或文档,用向量矩阵进行表示。矩阵的每一行表示个单元(token的向量(vectors,通常是个单词的词向量(word embeddings),可用word2vec或Glove -)II练得到。个句子或一篇文档的各个单词进行纵向排列,构成了矩阵的列。下图2显示了NLP句子分类任务的经典CNNs模型。
2.3.2 CNNs超参数
CNNs运行中有一些重要的超参数 ( hyperparameters)需要确定,主要包括: (1)窄卷积与宽卷积(Narrow vs. wideoonvolution:对句子进行补零操作(zero-padding)称为宽卷积,不使用补零是窄卷积。进行补零操作的目的是为了确保滤波器能应用到矩阵的每个元素,从而得到较大或大小一致的输出。(2)步长(Stridesize:表示滤波器每一次平移的距离,通常为1。(3)池化层(Pooling layer:是对输入值进行子采样,包括最大值池化(max pooling)和平均值池化(average pooling ) 。池化的作用提供固定输出大小的矩阵,为后续分类所需,同时降低输出的维度又保存了最重要的信息。(4)通道(Channels:是指输入数据的不同视角(views)。在图像识别中,有红绿蓝(Red, Green and Blue, RGB通道。在NLP中,有不同词表示的通道,如word2vec和G1oVe,或同句子以不同语种表示或以不同方式进行解析。
2.3.3 代表性CNNs研究
Kim (2014)报道了将CNNs引入文本和关系分类的开创性土作,提出用于NLP处理的经典CNNs框架,其组成为:(1)输入层,是用词向量表示句子的矩阵(n * k),n表示句子长度补齐后的总单词数,k表示单词的词向量的维度;(2)卷积层,用多个不同宽度的滤波器,并得到特征映射 (feature maps);(3)最大池化层;(4)全连接层,进行dropout操作、用softmax多类分类器得到分类结果。在不同分类数据集用此架构进行分类,方法取得不错的效果。Zeng等(2014)对输入信息进行了丰富,包括词汇级(lexical level和句子级(sentence level)特征。其中,句子级特征将表示句子的词向量与两个实体的位置向量作为输入,投入CNNs框架,最后融合所有特征进行分类,取得显著的效果提升。为解决短文本分类中数据稀疏和歧义性问题,Wang ( 2015等将语义聚类结果作为输入投入CNNS。通过快速聚类算法在向量空间(embedding spaces)发现语义子团(semantic cliques,并进步遴选出有意义的语义单元(semantic units,再投入到卷积层中。在短文本分类的两个开放评测中,结果验证了方法的有效性。Chen (2015)等在情感分类中融合情感词典和CNNs,在COAE2014微博数据集上有效地提升情感分类的效果。从前述研究中可看到,增强输入内容的语义相关性、提升CNNS架构的自身表示能力,是CNNs在公共领域优化关系分类效果的努力方向。CNNs部分代表性研究内容请见表3o

在生物医学领域,Liu等(2016)报告基于CNNs的药物一药物相互作用(DDI)抽取,使用了词向量和位置向量。在2013 DDI关系抽取评测集合中取得F值69.75%,超过之前最佳结果2.75%o Zhao等(2016)]和Hua等(2016)均对训练语料进行最短依存路径(shortestdependency path, SDP)句法分析,旨在保留两个兴趣实体的最核心语境信息、去掉句子中不相关单词,并将SDP结果作为输入投入CNNs模型中,分别在DDI和PPI抽取中取得了良好效果。这几项最新研究表明,CNNs己从公共领域引入到生物医学领域,目一相比之前的研究取得了具有竟争性效果。鉴于生物医学语义关系的自动抽取方法有着紧迫的现实需求,深度学习为其带来了 一种全新的研究机制,值得进行进步探索与应用。
3 结论与展望
通过对生物医学SRE方法三个阶段 (基于知识、传统机器学习和深度学习)的系统分析,我们发现在前两个阶段,利用医学知识资源和NLP技术、构建合适的特征和选择恰当的核函数,是关系抽取的研究重点。在第三个阶段,以CNNs为代表的深度学习使用词向量来表示单词,从手土定义的复杂特征和规则中解放出来,并减少了对自然语言处理的依赖,实现了良好的效果。然而,CNNs基于词向量的输入机制也如同 一把双刃剑:一方面,它避免了自然语言处理过程中产生的错误,也无需预定义复杂的特征;而另一方面,它不易整合外部资源,而后者很可能对其提升效果大有裨益,特别是在既有充足知识资源又对精准度要求很高的医学领域。
基于前述调研,生物医学SRE可从以下方面做进步的探索。
(1)利用深度学习方法提升效果。由于深度学习的模型构建和参数训练比较复杂,对系统计算性能有较高要求,目前关系抽取的深度学习方法主要由计算机科学家在开放领域进行研究。在医学领域,深度学习更多集中在医学影像处理(如对CT、核磁共振等图像结果进行模式识别与辅助诊断),而对医学文本处理的报道较少。IBM沃森(Watson智能系统通过自动阅读大量文献和临床病历信息,能提出具有参考价值的诊疗意见,为发挥医学文本处理的宝贵价值起到了良好的引领作用。令人欣喜的是,深度学习社区提供了大量开源代码和不同层级的框架(如Keras, TensorFlow,减少了编码要求、降低了学习门槛,为生物医学SRE研究搭上深度学习的快车提供了便利。因此,彻底理解各种深度学习模型的原理和优缺点,加以恰当使用,可望在新研究范式下提升生物医学SRE的自动化水平。
(2)增强与先验知识的融合。目前深度学习方法主要是基于表示学习(将单词转化为语义向量)机制作为输入层,不易与显式的符号自然语言融合,因此忽略对知识资源的利用。研究表明,对任务进行更深度的分析、更好整合先验知识,实现数据与知识的双重驱动,将会促进研究结果的质量和效果提升。因此,在拥有丰富研究历程的生物医学领域,融合先验知识(如医学知识组织体系、己证实的重要特征等)到深度学习模型中,优化输入信息和框架结构以提升语义表示能力,将是个充满前景的探索方向。
(3)加强语料库的建设。由于生物医学事关生命与健康,其重要实体(疾病、药物、基因等)之间的关系构建需要保障高的精准度。相对于无监督学习,有监督学习有更好的准确性和稳定性,而良好构建的语料库是有监督方法得以开展的关键前提。目前,由公开测评提供的标注语料,常常为百篇或千篇级文本,数据规模偏小,难以充分支持深度学习的训练。在图像识别领域,数据量大的ImageNet(十亿多张图片)、语义标识丰富的Visual Genome(十余万张图片)等数据库,为深度学习在计算机视觉的成功探索发挥了巨大作用。生物医学领域一直重视基础设施建设,如一体化医学语言系统(UMLS)的超级叙词表 ( Metathesaurus整合百余部词表和百万级的规范概念,NCBO BioPortal本体库包含数百个不同来源的生物医学本体。在人工智能时代,充分利用医学基础设施建设的成功经验和优良传统,借助众包、机构协作等各种形式,建设高质量和大数据量的语料集,为机器学习提供重要支撑。
(4)加强领域内外合作。生物医学领域存在较高的知识壁垒,积累了大量知识资源,因此,计算机科学家、NLP专家与生物医学专业人员紧密合作有助于更好地理解问题、利用资源,使生物医学SRE获得更好的研究效果,发挥更大的实用价值。


 362716231
362716231
